Mutagenesi
Enciclopedia on line
In genetica, l’insieme dei processi chimico-fisici che portano a una mutazione. Gli agenti mutageni sono composti chimici (m. chimica) e radiazioni (m. fisica).
La classe dei mutageni fisici è costituita dalle radiazioni ionizzanti (raggi X, raggi γ, particelle α e β ecc.), dalle radiazioni ultraviolette e dalla temperatura. I raggi X, fondamentalmente, producono rotture a un singolo o ad ambedue i filamenti di DNA e legami crociati (cross-links) tra varie molecole di DNA o tra DNA e proteine: gli ultravioletti producono dimeri di timina e denaturazione del DNA nella regione dove si forma il dimero.
La classe dei mutageni chimici, invece, è molto più ampia e include sostanze varie che possono, però, essere raccolte in base al loro meccanismo di azione: a) sostanze che si sostituiscono alle basi del DNA (come gli analoghi delle basi 5-bromouracile e 2-amminopurina); b) sostanze in grado di reagire con le basi del DNA (come acido nitroso, idrossilammina, etil-etansolfonato e iprite); c) sostanze che si intercalano fra le basi del DNA (come le acridine).
La popolazione umana è comunque sottoposta ogni giorno a un certo numero di sostanze che possono essere mutagene: per es., circa 70.000 specie chimiche sintetiche sono usate nel commercio e un terzo di esse sono relativamente comuni. È possibile che si verifichi un danno genetico ingente sia alle cellule germinali con conseguente nascita di individui malformati sia, soprattutto, alle cellule somatiche con conseguente insorgenza di tumori, senza che si possa essere sempre in grado di associare un mutageno con il suo effetto. Nell’uomo un potenziale mutageno ingerito passa attraverso le varie modificazioni che avvengono nella bocca, nello stomaco, nell’intestino e soprattutto nel fegato, dove la sostanza può essere accumulata o trasformata dagli enzimi epatici. Molti mutageni sono pertanto neutralizzati dai vari sistemi enzimatici che incontrano, ma altre sostanze possono diventare mutagene proprio in seguito ai processi metabolici che subiscono.
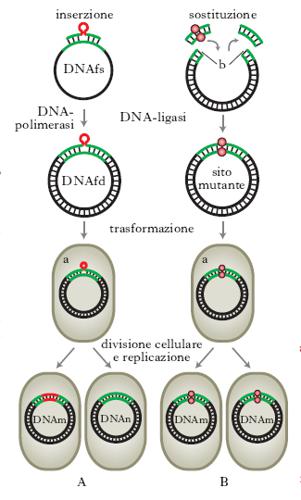
Per studiare la funzione dei geni, i genetisti utilizzano tradizionalmente cellule e organismi mutati in qualche funzione. Usando cellule od organismi che si riproducono rapidamente non è infatti difficile ottenere sperimentalmente grandi quantità di mutanti, anche se gli studi che occorrono successivamente per associare la mutazione di un gene all’alterazione di una particolare proteina possono essere invece complessi e richiedere un tempo molto lungo. La tecnologia del DNA ricombinante (➔ biotecnologie) ha reso possibile un nuovo approccio sperimentale, talvolta chiamato genetica inversa, dato che inverte la direzione della tradizionale analisi genetica, o anche, più correttamente, clonazione posizionale. Le tecnologie sulle quali si basa questo approccio sperimentale permettono di identificare e clonare direttamente un gene, di alterarlo in siti programmati e, successivamente, di identificare la proteina da esso codificata. Qualsiasi frammento di DNA proveniente da qualsivoglia organismo può essere inserito in un vettore molecolare batterico e poi clonato. Questo inserto, derivato sia da Procarioti sia da Eucarioti, può essere modificato in un punto preciso per m. diretta e reintrodotto nei batteri, nelle piante o nei Vertebrati che cambieranno così le loro caratteristiche. Per introdurre nel DNA mutazioni di singole basi, si usa comunemente una tecnica, detta m. sito-specifica: il primo passo consiste nella sintesi chimica di un oligonucleotide nel quale vengono introdotti i cambiamenti di basi desiderati. L’oligonucleotide sintetico, che contiene la porzione alterata della sequenza nucleotidica del gene, viene ibridato con la sequenza del gene non mutato che è stato inserito in un vettore a singolo filamento (v. fig.). Le condizioni di ibridazione devono permettere l’appaiamento anche se i due filamenti non sono perfettamente complementari. Successivamente l’oligonucleotide sintetico servirà da innesco per la sintesi in vitro della doppia elica di DNA del vettore che presenterà la sequenza alterata in uno dei due filamenti. Il vettore, inserito poi in Escherichia coli, si replicherà e formerà parecchie copie di DNA mutato su entrambi i filamenti. Il metodo della m. sito-specifica può tuttavia essere usato anche su geni clonati in vettori a doppio filamento, a condizione che il DNA sia prima denaturato.
È inoltre possibile modificare opportunamente un gene clonato con un metodo chiamato sostituzione a cassetta: si rimuove una parte del gene mediante due tagli con enzimi di restrizione (➔ enzima) e si sostituisce il frammento (cassetta) con un altro variamente modificato. Alla fine degli anni 1990 è stata messa a punto per la m. una procedura che utilizza la PCR. Un primer contenente la mutazione viene usato per un primo ciclo di PCR; il prodotto di questo primo ciclo viene poi usato come innesco per un secondo ciclo, che dà come prodotto il gene mutato. Il gene con la mutazione desiderata può essere trasferito in una cellula dove può diventare parte permanente del suo genoma, replicarsi con esso e codificare la proteina alterata della quale si vuole analizzare la funzione; mediante la m. diretta e mirata si può capire, per es., quali parti della catena polipeptidica di un enzima sono importanti nei processi fondamentali di ripiegamento, di interazione con il ligande e di catalisi.
Se il gene si trasferisce invece in un uovo fecondato o in cellule staminali embrionali si otterranno interi organismi multicellulari mutati in grado di trasmettere la mutazione alla loro progenie come parte permanente della propria linea germinale. Questi animali, detti transgenici, costituiscono (così come quelli knock out) utili modelli sperimentali per lo studio della funzione e della regolazione dei geni in vivo e, in modo particolare, per lo studio di quelle mutazioni che determinano malattie ereditarie nell’uomo.